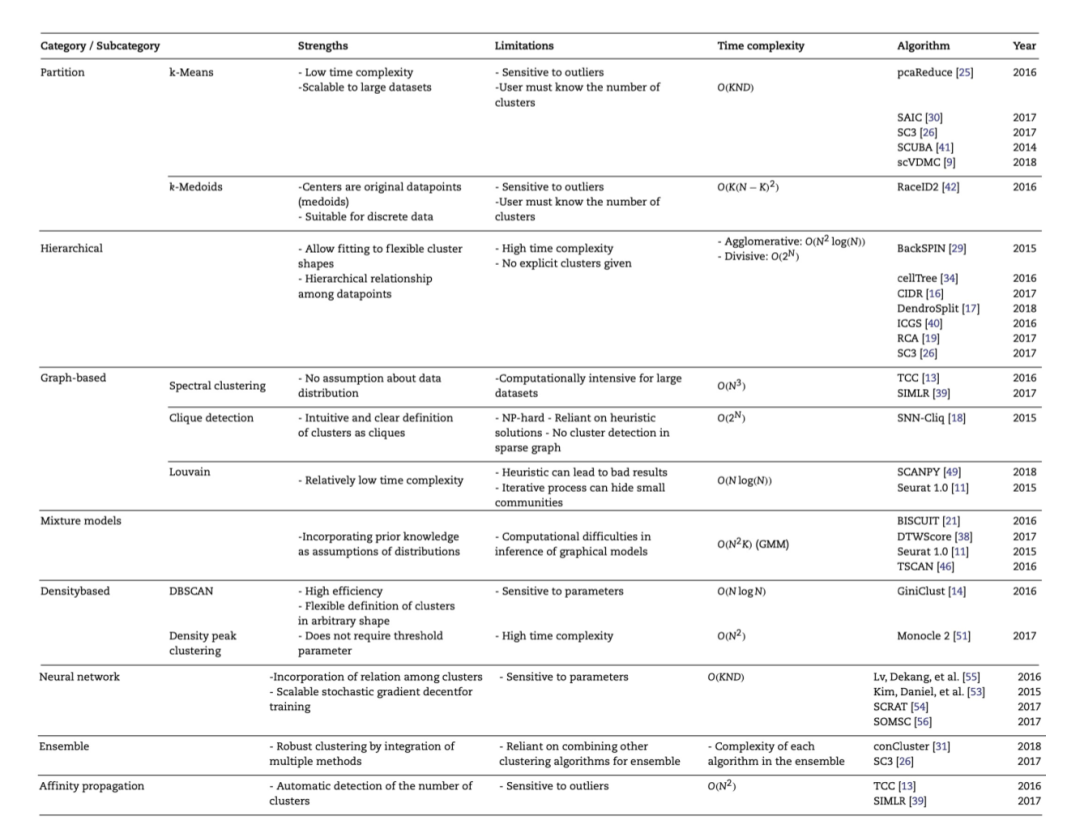
在本節中,我們將闡述八種應用在單細胞轉錄組數據的聚類方法,并總結了這些方法的優點、局限性和時間復雜度等。一部分單細胞聚類的工具會使用多種聚類算法,因此會在多個類別中列出。
01
基于劃分的聚類
基于劃分的聚類方法主要是確定最佳的K個中心,將數據點劃分為K個簇,其中心要么是質心(均值),稱為k-means,要么是中心點,稱為k-medoids。 k-means方法的思想是找到質心,以最小化每個數據點與其最近質心之間的歐氏距離的平方和。它具有時間復雜度低的優點。但是,它對異常值很敏感,并且用戶必須預先指定聚類的數量K。對于將N個D維數據點聚為K個類,使用Lloyd 's算法的k-means每次迭代的時間復雜度為O(KND)。 以下是一些使用k-means聚類的單細胞轉錄組數據分析工具。SAIC在迭代聚類過程中使用k-means并結合ANOVA識別特征基因;SCUBA使用k-means將每個時間點的細胞分為兩組,并使用間隔統計量來識別分叉事件;SC3的步驟之一是在細胞距離矩陣上使用k-means聚類(圖3)。k-medoids方法是將原始N個數據點中的K個數據點識別為中心點,以最小化數據點到中心點的距離之和。它非常適用于以有意義的中心點作為聚類中心的離散數據。然而,與k-means類似,它對異常值很敏感,用戶必須預先指定聚類的數量K。對于從N個數據點中選擇最優K個點的組合問題,采用圍繞中心點劃分算法的k-medoids的時間復雜度為O(K(N?K)2)。
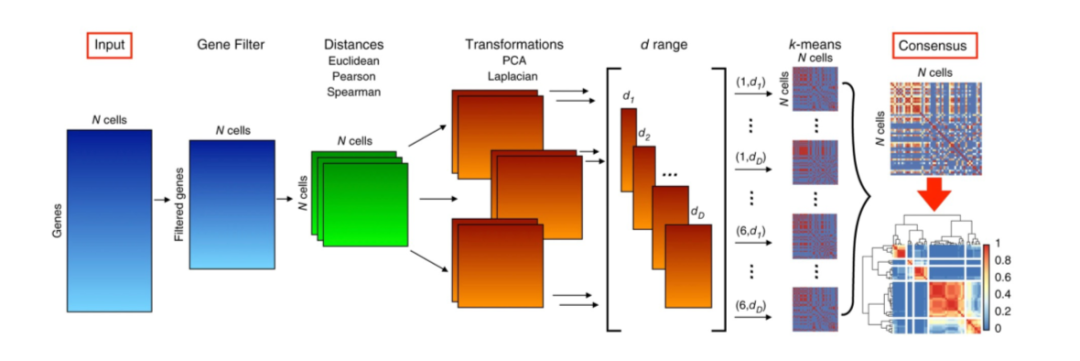
Figure 3. SC3聚類流程圖
RaceID2用于利用單細胞轉錄組數據識別罕見細胞類型,結果表明用k-medoids取代k-means方法進行聚類可以明顯改善聚類的結果。
02
層次聚類
次聚類是基因表達數據分析中應用最廣泛的聚類方法。層次聚類在數據點之間構建層次結構,它根據層次樹中的分支定義不同的類群。許多單細胞轉錄組數據的聚類算法都是基于層次聚類或將層次聚類作為分析的步驟之一。
層次聚類對數據本身的分布并沒有過多要求,因此,它適用于許多不同形狀的數據集;層次聚類的另一個優點是通過數據點之間的層次關系,可以用于發現其內在的關聯,有助于對結果的解釋。層次聚類主要有兩種實現方法:聚合式(agglomerative)和分裂式(divisive)。 聚合式又叫“自下而上式(bottom-up)”的聚類,它從N個數據點開始,每一個數據點作為一個單獨的類,在每一步中,類群依據它們之間的距離進行合并,直到所有類群在層次結構的根處合并在一起。分裂式又叫“自上而下式(top-down)”聚類,相比之下,該方法首先將所有數據點當成一個類群,然后每一步遞歸劃分更小的類群,直到分成N個類群為止。無論是哪一種,層次聚類的一個顯著缺點是時間復雜度高,運行時間非常久。此外,層次關系并不能提供數據點的最佳聚類劃分,還需要一個額外的步驟來從層次樹中決定最終劃分的類群數量。 BackSPIN是一種雙聚類算法,分別在細胞和基因的維度上應用層次聚類。BackSPIN使用SPIN迭代地拆分基因表達矩陣,直到在分支處不再滿足拆分標準;cellTree通過在話題分布上構造最小生成樹,從而在單個細胞之間構建層次結構;CIDR對PCoA獲得的低維嵌入使用了層次聚類;ICGS采用層次聚類,將篩選后得到的一組基因的表達數據按表達水平和動態范圍進行聚類,并進行配對相關分析;SC3對多個k-means聚類結果合并得到的一致性矩陣進行層次聚類;為了獲得層次結構中的實際類群,DendroSplit通過衡量與原始表達數據的分離分數,使用動態拆分和合并分支來檢測層次樹中的類群。
03
混合模型
混合模型聚類基于的假設思想是,數據點是從幾個混合的概率分布中采樣,每個概率分布代表一個聚類。樣本的聚類是通過從每個分布中學習其生成的概率來推斷的。用于聚類的常見混合模型主要包括應用于連續型數據的高斯混合模型(GMM)和計數型數據的分類混合模型。
混合模型的優點包括嚴格的概率建模和在模型中引入先驗知識的靈活性。然而,解決混合模型需要先進的優化或采樣技術,具有較高的計算復雜度,并依賴于關于數據分布的假設的準確性。混合模型通常是用期望最大算法學習的,它可以推斷混合參數和類分配似然性,也可以用抽樣和變分方法學習圖概率模型。此外,混合模型的時間復雜度取決于混合的分布,比如在GMM中,時間復雜度為O(N2K)。 BISCUIT基于層次狄利克雷過程混合模型(HDMM),并附加細胞特定的標準化和dropouts矯正。它的過程首先是應用HDMM對細胞建模,形成包含Dirichlet先驗的混合系數、均值、Wishart先驗的協方差矩陣的高斯混合模型,而細胞特定的縮放因子代表了技術變異。早先版本的Seurat能夠將單細胞轉錄組數據與原位RNA測序相結合,用于單細胞的空間聚類。在雙峰混合模型中,針對一組選定的標志基因,將單細胞轉錄組數據與二值化的原位RNA數據整合,然后通過雙峰混合模型中單細胞轉錄組表達譜的后驗概率將每個單細胞分配到不同的空間類群區域。
04
基于圖的聚類
在基于圖的聚類中,數據點被表示為圖(Graph)中的節點,而節點間的邊(Edge)由數據點之間的相似性表示。基于圖的聚類基于一個簡單的假設,即圖中的密集社區(community)表示為密集的子圖或譜成分,因此對于數據的分布并沒有過于依賴。兩種最常用的圖聚類算法是譜聚類和團(clique)發現。
在譜聚類中,通過相似函數(如RBF核函數)建立相似性矩陣及其拉普拉斯圖。通過計算拉普拉斯圖的頂部特征向量,以便后續的k-means聚類。雖然可以使用更有效的方法來尋找固定數量的頂部特征向量,但尋找所有特征向量的時間復雜度為O(N3),因此,譜聚類并不適用于大數據集。當細胞類型作為先驗已知時,基于TCC的聚類利用細胞間的Jensen-Shannon距離構建相似性矩陣進行譜聚類;未知時則應用近鄰傳播聚類。 在圖論中,團被定義為每對節點都相鄰的子圖,因此,團代表了圖中數據點的類群。由于在圖中找到團是一個NP-hard問題,通常會使用啟發式方法。SNN-Cliq利用單細胞轉錄組數據對細胞進行團簇檢測。在稀疏圖中團通常很少見,因此,SNN-cliq在SNN圖中檢測到的團一般是密集但是不完全連通的。
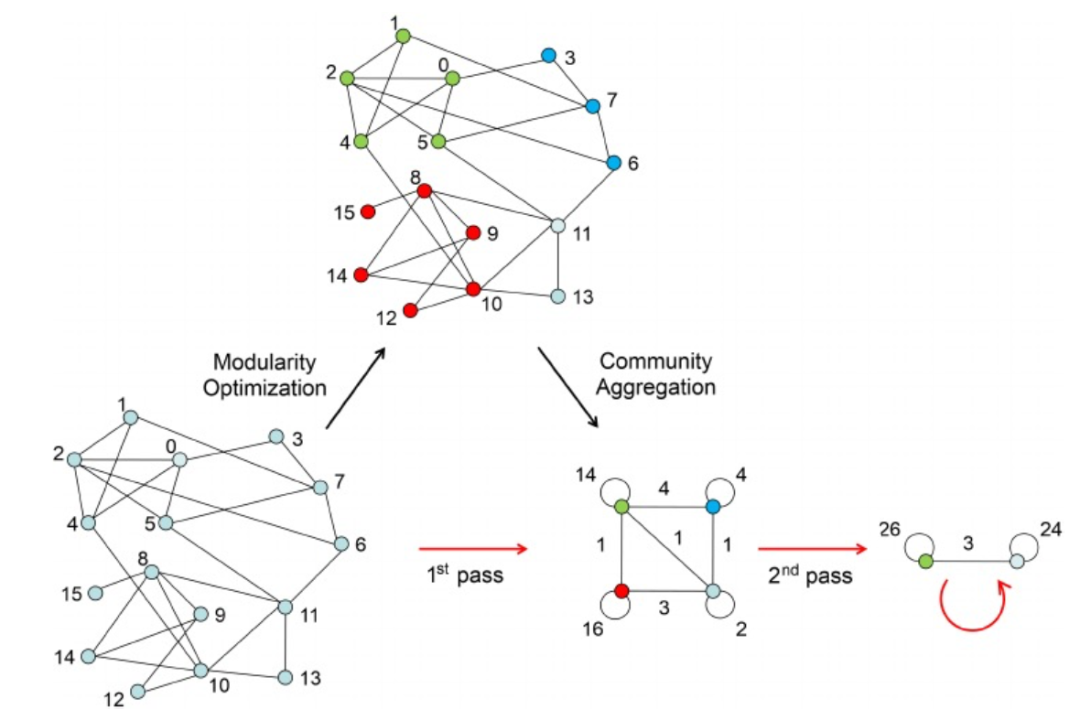
Figure 4. Louvain算法示意圖
另一種常用于單細胞分析的基于圖聚類的算法是Louvain算法(圖4)。Louvain是一種社區檢測算法,它比其他基于圖的算法更具可擴展性,通過貪婪方式將節點分配給社區,并更新網絡以獲得低分辨率的聚類。Louvain的時間復雜度為O(NlogN)。Scanpy是一個集成了Louvain算法、提供了一個能夠分析大規模單細胞轉錄組數據集的工具。Seurat在默認情況下也是利用Louvain算法在細胞SNN圖上來發現細胞類型。
05
基于密度的聚類
基于密度的聚類將類群定義為一個空間中具有高密度數據點的區域。DBSCAN和密度峰值聚類是基于密度的聚類的兩個例子。
給定一個數據點,將其作為中心以ε為半徑劃分出一個球形,球形內的數據點數量如果超過指定的閾值,那么這些數據點就被DBSCAN認為是一個類群。對每個數據點重復該過程,不斷擴展,最終完成聚類。該方法具有效率高、適用于任何形狀的數據的優點。然而,密度聚類對參數非常敏感,如果類群密度不平衡,結果會非常差。DBSCAN聚類的時間復雜度為O(NlogN)。基于密度的聚類通常用于單細胞轉錄組數據分析中的異常細胞識別,如GiniClust和Monocle2。 GiniClust是基于DBSCAN來發現罕見的細胞亞群,它使用基尼指數作為基因表達值變異性的衡量標準,以篩選高變基因,然后由DBSCAN對細胞聚類。密度峰值聚類考慮數據點之間的距離,而不是像DBSCAN那樣考慮密度閾值,同時假設聚類的中心是聚類中數據點密度的局部最大值。密度峰值聚類的時間復雜度為O(N2)。在Monocle2中,就是對t-SNE空間內的細胞進行密度峰值聚類。
06
Kohonen神經網絡,也稱為自組織特征映射神經網絡(SOMs),運用競爭學習策略逐步優化網絡進行聚類,使用隨機梯度下降通過不斷迭代訓練數據點和每個中心的相似度來更新聚類中心。類群中心使用預定義的結構(如網格)進行初始化。SOM具有相當強的可擴展性,因為隨機梯度下降不需要把所有的數據點保存在計算機內存中。此外,中心之間的預定義結構可以引入先驗知識,并在類群之間提供可解釋的關系。然而,SOM對參數異常敏感,比如用于更新權重的學習率。
SOM也已用于單細胞轉錄組數據的可視化和聚類。在一些研究中應用SOM在二維熱圖中直觀的可視化相似關系。SCRAT為用戶提供了可視化二維熱圖的選項,該熱圖反映了跨細胞群的基因之間的相關性。SOMSC利用SOM將高維基因表達數據折疊成二維,用于識別處于中間過渡狀態的細胞以及擬時間排序。
07
集成聚類
集成聚類,也稱為共識聚類,是一種廣泛使用的策略。在該策略中,通過不同的應用場景(例如不同的聚類算法,相似的度量和特征選擇/映射等)對同一數據集進行聚類,然后基于單個聚類結果之間的一致性,通過共識函數對它們進行合并。集成學習可以捕獲不同數據或聚類模型中的多樣性,并且已被證明比單一模型更健壯,并產生更好的結果。集成聚類的局限性是依賴于其他的數據轉換和基本聚類方法。
SC3是一種用于單細胞轉錄組數據聚類的共識聚類方法。SC3首先通過三種不同的度量(斯皮爾曼、皮爾森和歐氏距離)來計算細胞間相似性,然后使用PCA和拉普拉斯轉換進行分解,通過k-means對不同類型的低維嵌入進行聚類,接著用CSPA共識函數構建一致性矩陣,最后,利用該矩陣進行層次聚類。conCluster是另一種共識聚類方法,它使用多個不同的參數通過t-SNE和k-means進行了組合,然后將這些不同的組合連接起來,用于最后的k-means聚類。
08
近鄰傳播聚類
該聚類方法的主要思想是通過不同點之間的信息傳遞來選擇聚類中心:吸引度(responsibility)用于描述一個數據點k作為數據點i的聚類中心的適合程度;歸屬度(availability)則描述了數據點i選擇數據點k作為聚類中心的適合程度。近鄰傳播聚類的主要優點是不需要知道類群的數量。缺點是時間復雜度較高,對異常值敏感。當細胞類型數量未知時,基于TCC的聚類以該方式進行細胞的聚類。在SIMLR中也選項可以選擇對數據進行該方法的聚類。
Table 1聚類方法的分類及優缺點
審核編輯 :李倩
-
神經網絡
+關注
關注
42文章
4812瀏覽量
103215 -
算法
+關注
關注
23文章
4705瀏覽量
95094 -
聚類
+關注
關注
0文章
146瀏覽量
14390 -
機器學習
+關注
關注
66文章
8499瀏覽量
134331
原文標題:單細胞轉錄組 | 聚類分析中的機器學習與統計方法綜述(二)
文章出處:【微信號:SBCNECB,微信公眾號:上海生物芯片】歡迎添加關注!文章轉載請注明出處。
發布評論請先 登錄





 聚類分析中的機器學習與統計方法綜述(二)
聚類分析中的機器學習與統計方法綜述(二)













評論