單細胞測量技術發展迅速,徹底改變了生命科學。我們已經從測量幾十個細胞擴展到數百萬個細胞,從一種模式擴展到多個高維模式。單個細胞水平上的大量信息為訓練機器學習模型提供了一個很好的機會,幫助我們更好地理解 intrinsic link of cell modalities ,這可能會對合成生物學和 drug target discovery 產生變革。
這篇文章介紹了模態預測,并解釋了我們如何用基于 NVIDIA GPU 的 RAPIDS cuML 實現取代基于 CPU 的 TSVD 和內核嶺回歸( KRR ),從而加速了 NeurIPS Single-Cell Multi-Modality Prediction Challenge 的獲勝解決方案。
使用 cuML ,只修改了六行代碼,我們加速了基于 scikit 學習的獲勝解決方案,將訓練時間從 69 分鐘縮短到 40 秒:速度提高了 103.5 倍!即使與 PyTorch 中開發的復雜深度學習模型相比,我們觀察到 cuML 解決方案對于這種預測挑戰來說更快更準確。
多虧了單細胞技術,我們可以測量同一單細胞內的多種形態,如 DNA 可達性( ATAC )、 mRNA 基因表達( GEX )和蛋白質豐度( ADT )。圖 1 顯示了這些模式之間的內在聯系。只有可獲得的 DNA 才能產生 mRNA ,而 mRNA 又被用作生產蛋白質的模板。
當期望從一個模態預測另一個模態時,模態預測的問題自然出現。在 2021 NeurIPS challenge 中,我們被要求預測從 ATAC 到 GEX 以及從 GEX 到 ADT 的信息流。
若一個機器學習模型能夠做出好的預測,那個么它一定已經了解了細胞的復雜狀態,它可以為細胞生物學提供更深入的見解。擴展我們對這些調控過程的理解,對于藥物靶點的發現也具有革命性意義。
模態預測是一個多輸出回歸問題,它提出了獨特的挑戰:
High cardinality.例如, GEX 和 ADT 信息分別以長度為 13953 和 134 的矢量描述。
Strong bias.數據收集自 10 個不同的捐贈者和 4 個地點。培訓和測試數據來自不同的站點。捐贈者和站點都強烈影響數據的分布。
稀疏、冗余和非線性。模態數據稀疏,列高度相關。
在這篇文章中,我們專注于 GEX 到 ADT 預測的任務,以證明單個 – GPU 解決方案的效率。我們的方法可以擴展到使用多節點多 GPU 架構的具有更大數據量和更高基數的其他單細胞模態預測任務。
使用 TSVD 和 KRR 算法進行多目標回歸
作為基線,我們使用了密歇根大學鄧凱文教授的 NeurIPS 模態預測挑戰“ GEX 到 ADT ”的 first-place solution 。核心模型的工作流程如圖 2 所示。訓練數據包括 GEX 和 ADT 信息,而測試數據只有 GEX 信息。
任務是預測給定 GEX 的測試數據的 ADT 。為了解決數據的稀疏性和冗余性,我們應用截斷奇異值分解( TSVD )來降低 GEX 和 ADT 的維數。
特別是,兩種 TSVD 模型分別適用于 GEX 和 ADT :
對于 GEX , TSVD 適合訓練和測試的級聯數據。
對于 ADT , TSVD 僅適合訓練數據。
在鄧的解決方案中, GEX 的維度從 13953 大幅降低到 300 , ADT 從 134 大幅降低到 70 。
主成分 300 和 70 的數量是通過交叉驗證和調整獲得的模型的超參數。然后將訓練數據的 GEX 和 ADT 的簡化版本與 RBF 核一起饋送到 KRR 中。根據鄧的方法,在推理時,我們使用經過訓練的 KRR 模型執行以下任務:
預測測試數據的 ADT 的簡化版本。
應用 TSVD 的逆變換。
恢復測試數據的 ADT 預測。
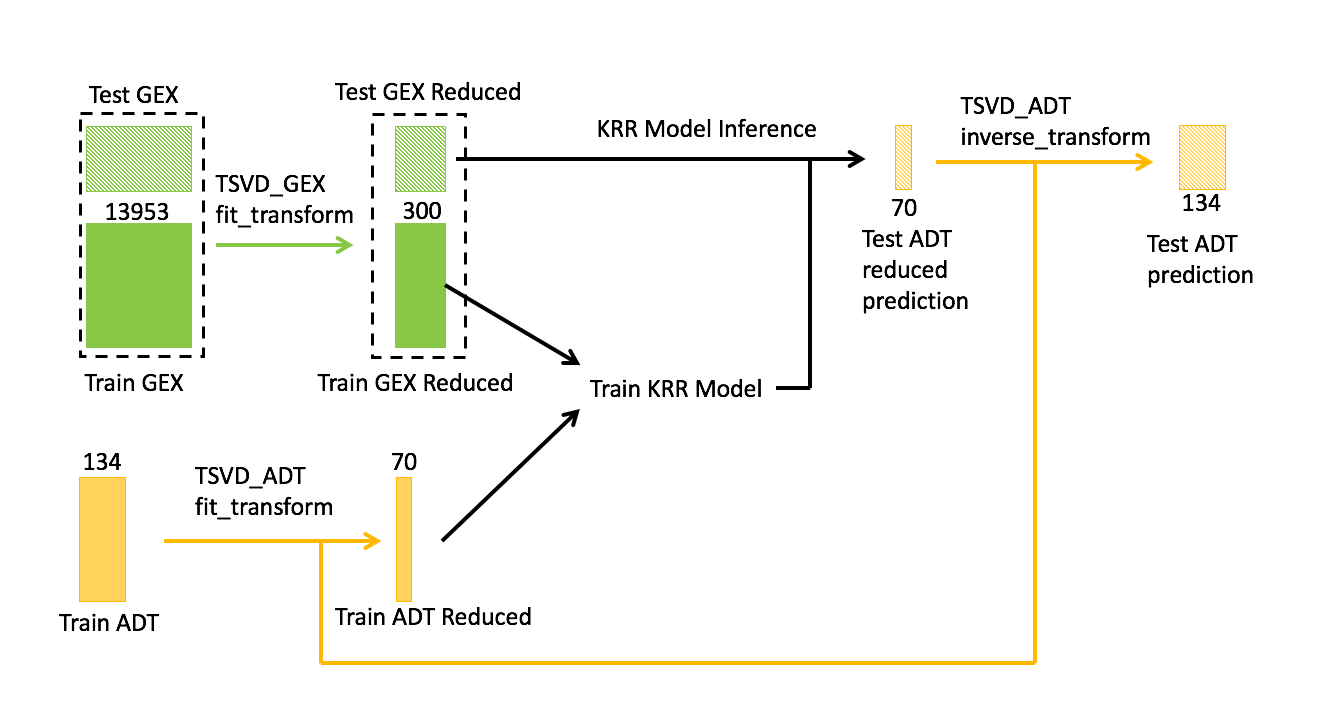
圖 2.模型概述。塊表示輸入和輸出數據,塊旁邊的數字表示尺寸。
通常, TSVD 是對稀疏數據執行降維的最常用選擇,通常在特征工程期間使用。在這種情況下, TSVD 用于減少特征( GEX )和目標( ADT )的尺寸。目標的降維使下游多輸出回歸模型更加容易,因為 TSVD 輸出在列之間更加獨立。
選擇 KRR 作為多輸出回歸模型。與 SVM 相比, KRR 同時計算輸出的所有列,而 SVM 一次預測一列,因此 KRR 可以像 SVM 一樣學習非線性,但速度更快。
使用 cuML 實現 GPU 加速解決方案
cuML 是 RAPIDS 庫之一。它包含一套 GPU 加速機器學習算法,可提供許多高度優化的模型,包括 TSVD 和 KRR 。您可以將基線模型從 scikit 學習實現快速調整為 cuML 實現。
I在下面的代碼示例中,我們只需要更改六行代碼,其中三行是導入。為簡單起見,省略了許多預處理和實用程序代碼。
Baseline sklearn implementation:
from sklearn.decomposition import TruncatedSVD from sklearn.gaussian_process.kernels import RBF from sklearn.kernel_ridge import KernelRidge tsvd_gex = TruncatedSVD(n_components=300) tsvd_adt = TruncatedSVD(n_components=70) gex_train_test = tsvd_gex.fit_transform(gex_train_test) gex_train, gex_test = split(get_train_test) adt_train = tsvd_adt.fit_transform(adt_train) adt_comp = tsvd_adt.components_ y_pred = 0 for seed in seeds: gex_tr,_,adt_tr,_=train_test_split(gex_train, adt_train, train_size=0.5, random_state=seed) kernel = RBF(length_scale = scale) krr = KernelRidge(alpha=alpha, kernel=kernel) krr.fit(gex_tr, adt_tr) y_pred += (krr.predict(gex_test) @ adt_comp) y_pred /= len(seeds)
RAPIDS cuML implementation:
from cuml.decomposition import TruncatedSVD from cuml.kernel_ridge import KernelRidge import gctsvd_gex = TruncatedSVD(n_components=300) tsvd_adt = TruncatedSVD(n_components=70) gex_train_test = tsvd_gex.fit_transform(gex_train_test) gex_train, gex_test = split(get_train_test) adt_train = tsvd_adt.fit_transform(adt_train)adt_comp = tsvd_adt.components_.to_output('cupy')y_pred = 0 for seed in seeds: gex_tr,_,adt_tr,_=train_test_split(gex_train, adt_train, train_size=0.5, random_state=seed)krr = KernelRidge(alpha=alpha,kernel='rbf')krr.fit(gex_tr, adt_tr)gc.collect()y_pred += (krr.predict(gex_test) @ adt_comp) y_pred /= len(seeds)
cuML 內核的語法與 scikit learn 略有不同。我們沒有創建獨立的內核對象,而是在 KernelRidge 的構造函數中指定了內核類型。這是因為 cuML 還不支持高斯過程。
另一個區別是當前版本的 cuML 實現需要顯式垃圾收集。在這個特定的循環中創建了某種形式的引用循環,并且在沒有垃圾收集的情況下不會自動釋放對象。有關更多信息,請參閱 /daxiongshu/rapids_nips_blog GitHub 存儲庫中的完整筆記本。
后果
我們將 TSVD + KRR 的 cuML 實施與 CPU 基線和挑戰中的其他頂級解決方案進行了比較。 GPU 解決方案在單個 V100 GPU 上運行, CPU 解決方案在雙 20 核 Intel Xeon CPU 上運行。競爭的度量是均方根誤差( RMSE )。
我們發現, TSVD + KRR 的 cuML 實現比 CPU 基線快 103 倍,由于管道中的隨機性,分數略有下降。然而,比分仍然比比賽中的任何其他車型都好。
我們還將我們的解決方案與兩種深度學習模型進行了比較:
第四名解決方案:Multilayer Perceptron (MLP)
第二名解決方案:Graph Neural Network (GNN)
這兩個深度學習模型都在 PyTorch 中實現,并在單個 V100 GPU 上運行。這兩個深度學習模型都有許多層,需要訓練數百萬個參數,因此容易對該數據集進行過度擬合。相比之下, TSVD + KRR 只需訓練少于 30K 的參數。圖 4 顯示,由于其簡單性, cuML TSVD + KRR 模型比深度學習模型更快、更準確。
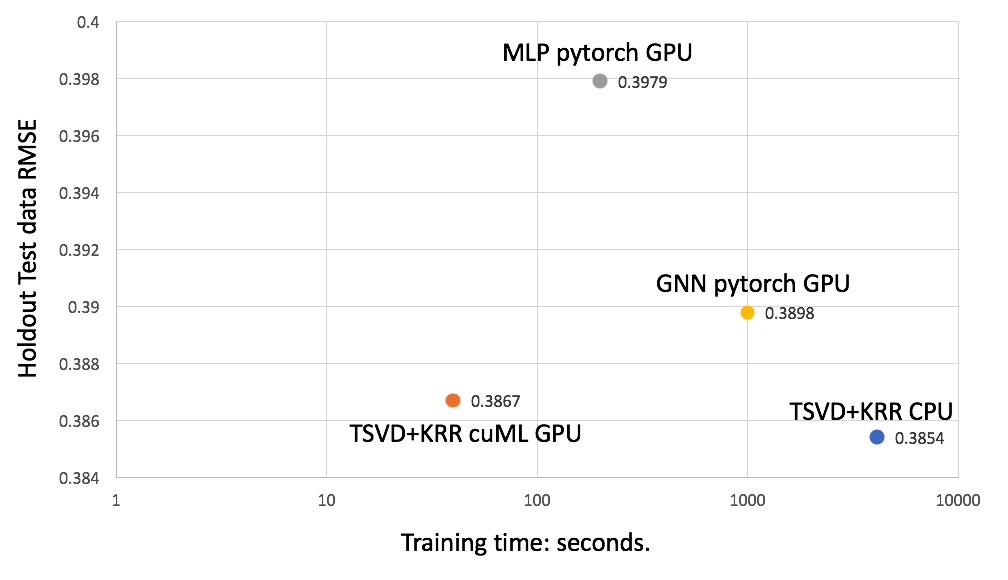
圖 4.績效和培訓時間對比。橫軸為對數刻度。
結論
由于其閃電般的速度和用戶友好的 API , RAPIDS cuML 對于加速單細胞數據的分析非常有用。通過少量的代碼更改,您可以提升現有的 scikit 學習工作流。
此外,在處理單細胞模態預測時,我們建議從 cuML TSVD 開始,以減少下游任務的數據維度和 KRR ,從而實現最佳加速。
使用 /daxiongshu/rapids_nips_blog GitHub 存儲庫上的代碼嘗試這個 RAPIDS cuML 實現。
-
NVIDIA
+關注
關注
14文章
5274瀏覽量
105922 -
AI
+關注
關注
88文章
34582瀏覽量
276128 -
電池
+關注
關注
84文章
11023瀏覽量
134305
發布評論請先 登錄
NVIDIA火熱招聘GPU高性能計算架構師
NVIDIA-SMI:監控GPU的絕佳起點
購買哪款Nvidia GPU
NVIDIA網格GPU-PSOD的支持結構
Nvidia GPU風扇和電源顯示ERR怎么解決
在Ubuntu上使用Nvidia GPU訓練模型
探求NVIDIA GPU極限性能的利器
Nvidia宣布推出了一套新的開源RAPIDS庫
GTC2022大會亮點:RAPIDS是NVIDIA備受歡迎的NVIDIA SDK之一

NVIDIA RAPIDS加速器可將工作分配集群中各節點
AutoML技術提高NVIDIA GPU和RAPIDS速度
RAPIDS cuDF將pandas提速近150倍






 如何用基于NVIDIA GPU的RAPIDS cuML實現取代KRR?
如何用基于NVIDIA GPU的RAPIDS cuML實現取代KRR?










評論