現有常用的空間轉錄組降維聚類方法,如主成分分析(principal component analysis,PCA)、非負矩陣分解(non-negative matrix factorization,NMF)等,多是基于單細胞數據進行開發的,因此不一定完全適用于空間轉錄組數據。例如,常用的降維聚類方法并沒有運用空間轉錄組所特有的組織空間定位信息,而僅僅只是基于表達譜進行聚類。但是,對于生物組織,相鄰的位置通常都具有相似的細胞組成和相似的基因表達水平。因此,將空間位置信息納入到降維聚類過程中,將更有利于實現接近生物實際狀態的空間聚類。
SpatialPCA就是基于以上觀點所開發的空間轉錄組降維聚類工具。該工具使用所謂的空間概率PCA(spatial probabilistic PCA),能夠明確地模擬組織位置間的空間相關性結構,在降維后的數據中保留原始空間數據的相鄰近似性。SpatialPCA將空間定位信息作為額外的輸入,使用核矩陣模擬組織位置間的空間結構。基于該方法所得到降維主成分因為包含相關的空間結構信息,稱其為空間主成分。具體來說,該方法同時使用基因表達矩陣和位置信息矩陣,將基因表達矩陣構建為一個潛在因子的函數模型。同時,根據位置信息矩陣對潛在因子構建核矩陣,以明確潛在因子的空間結構相關信息(圖1)。
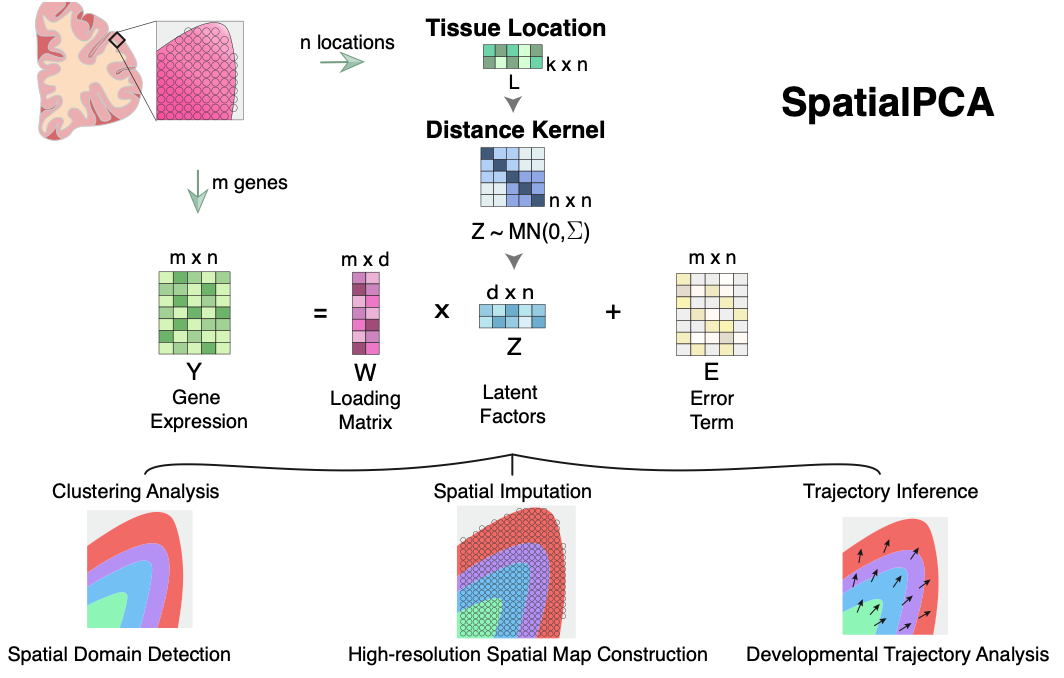
圖1
這里選取了背外側前額葉皮層(DLPFC)作為模擬數據的來源,通過手動分割各皮層,確定10000個特定細胞類型的細胞空間位置,并得到這些細胞的單細胞表達數據,設計了四種不同細胞組成情況,人工構建了四種空間轉錄組數據。相較于其他現有的空間聚類方法(包括采納空間信息與不采納空間信息的),SpatialPCA在模擬數據測試中體現出了更佳的檢測性能(圖2)。
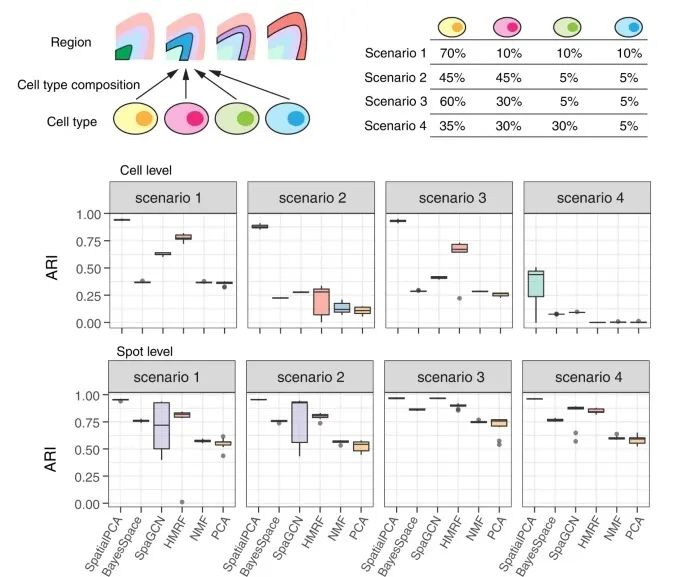
圖2
在真實數據的測試中,首先選取了人類DLPFC的Visium空間轉錄組數據,對多種工具(SpatialPCA, BayesSpace, SpaGCN, HMRF, stLearn, PCA, NMF)進行了平行測試。結果顯示,SpatialPCA生成空間聚類結果最接近實際的生物結構(圖3A),其預測的精確性和預測的空間域結構連續性(CHAOS打分越低連續性越高)也是幾種工具中最高的(圖3B)。
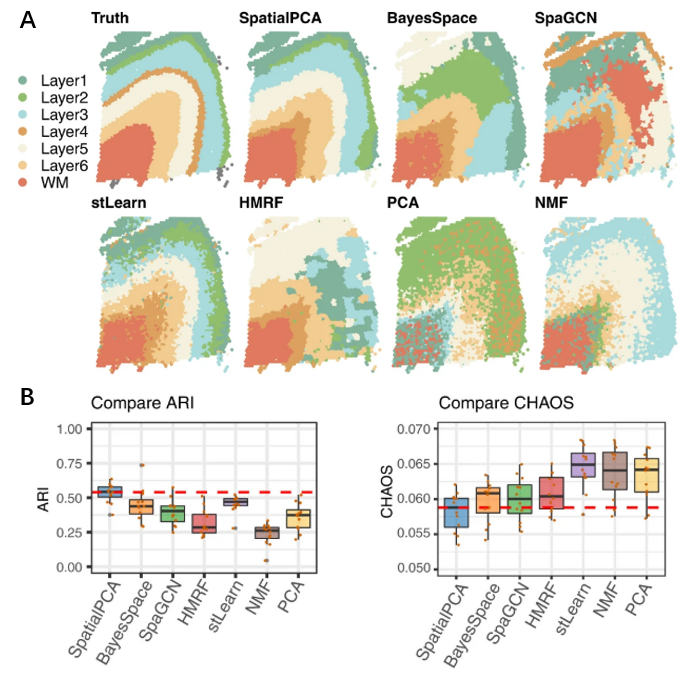
圖3
為了確認SpatialPCA的技術普適性,研究人員同時還在其他技術(Slide-seq,Slide-seqV2)產生的小鼠腦組織空間轉錄組數據中進行了測試。結果均顯示在多種工具中,SpatialPCA的聚類結果與實際情況更為符合(圖4)。而為了確認SpatialPCA的樣本普適性,利用HER2陽性乳腺腫瘤樣本進行測試,結果也顯示了SpatialPCA具有更好的聚類精確性和更符合實際情況。
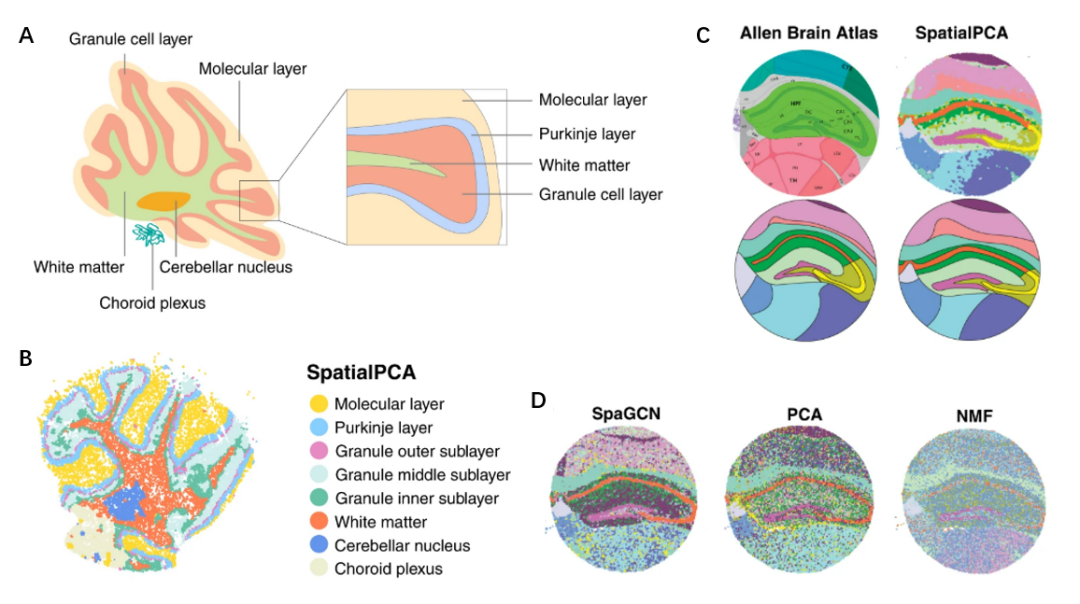
圖4
由于SpatialPCA所計算出的空間主成分同時包含了空間信息和基因表達信息,因此可以據此使用一些單細胞轉錄組分析工具來進行下游分析。通過使用Slingshot對腫瘤組織的空間轉錄組數據進行了軌跡推斷,顯示出了一條由腫瘤區域指向腫瘤周圍區域再到正常組織的軌跡(圖6A),計算得到的偽時間相關基因富集于免疫反應、細胞介導免疫和吞噬識別通路,凸顯了它們在癌癥進展、腫瘤侵襲和轉移中的重要性。
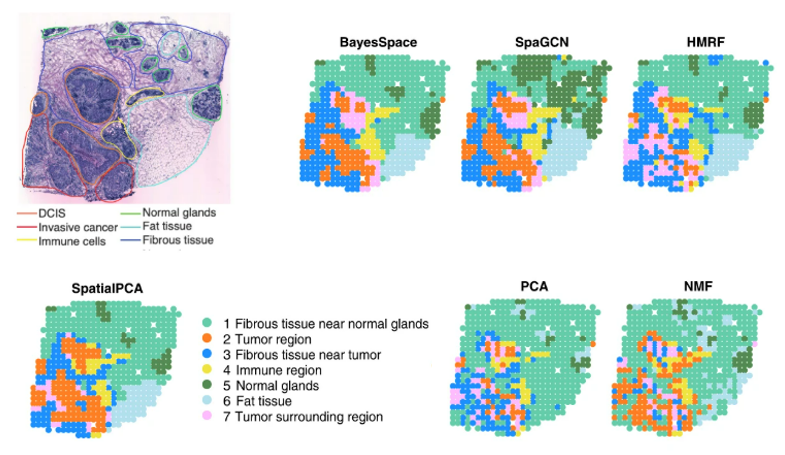
圖5
另外,根據SpatialPCA的建模框架,可以在新的空間位置上推斷基因表達水平,一次也可以通過低分辨率的樣本數據構建高分辨率的空間圖譜。對腫瘤組織的測試顯示,SpatialPCA所構建的高分辨率空間圖譜顯示出連續與平滑的特征,其聚類精確定位了不同組織區域間的邊界,并細化了緊鄰腫瘤和免疫區域之外的一層薄薄的腫瘤周圍區域(圖6B),顯示SpatialPCA在精細結構方面的優勢。
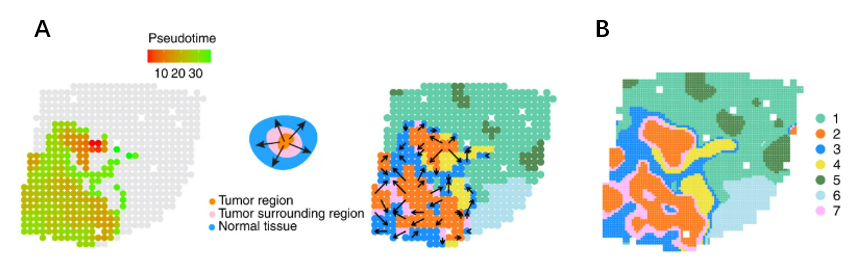
圖6
SpatialPCA是一種適合針對空間轉錄組數據降維聚類的分析工具,如果老師們手上的空間數據分群聚類不夠理想,趕快用SpatialPCA來試一試吧。
-
PCA
+關注
關注
0文章
91瀏覽量
30088 -
數據降維
+關注
關注
0文章
3瀏覽量
1296
原文標題:讓空間數據分析更簡單:數據降維聚類工具——SpatialPCA
文章出處:【微信號:SBCNECB,微信公眾號:上海生物芯片】歡迎添加關注!文章轉載請注明出處。
發布評論請先 登錄
云服務器計算池的運維團隊需要掌握的網絡工具
常見的三維測量方法和工具
福祿克儀表在數據中心運維中的應用
數據驅動AI工具在哪
PDA二維數據采集器的工作原理,二維碼采集器有什么推薦產品?

介紹6款開源免費的網絡監控工具






 數據降維聚類工具介紹——SpatialPCA
數據降維聚類工具介紹——SpatialPCA










評論