你去將你的基因組測序了嗎?世界上已有數百萬人去測過了,到2025年,這一數字可能會達到10億。
研究人員獲得的基因組數據越多,個人和公共健康的前景就越好。產前DNA測序已經可以篩查出發育異常。過不了多久,患者將可以對他們的血液進行測序,以發現任何可能標志著某種傳染病的非人類DNA。未來,與癌癥打交道的人將能夠通過每天對來自多個組織的細胞的DNA和RNA進行測序來跟蹤疾病的變化情況。
整個人群的DNA測序將使得我們可以對整個社會的健康狀況有更全面的了解。英國Biobank雄心勃勃,其目標是對50萬名志愿者的基因組進行測序,并跟蹤研究數十年。目前,人群范圍的基因組研究通常被用來識別與特定疾病相關的突變。定期對空氣、土壤和水中的生物進行測序將有助于追蹤流行病、食物病原體、毒素等等。
這樣的愿景的實現有賴于對超大量的數據的存儲和分析。通常情況下,DNA測序儀處理一個人的整個基因組就會產生數十至數百千兆字節的數據。數百萬人的基因組數據累加起來,所需要的存儲空間將達到數十艾字節。
而這僅僅是個開始。發現基因組數據有用的科學家、醫生和其他人不會僅對每個人進行一次測序——對于同一個個體,他們會希望隨著時間的推移對多個組織中的多個細胞進行重復測序。隨著測序速度的提高和成本的下降(現在個人基因組測序只需1000美元,而且價格正在快速下降),他們還希望對其他動物、植物、微生物和整個生態系統的DNA進行測序。而新應用甚至新產業的出現將帶來更多測序。
雖然很難預測基因組數據的全部未來收益,但我們已經看到了一個不可避免的挑戰:所需要的存儲空間幾乎是難以想象的大。目前,存儲基因組數據的費用仍然只是實驗室總體預算的一小部分。但是這種費用正在急劇升高,幅度遠遠超過了存儲硬件價格的下降。在未來五年內,存儲數十億人、動物、植物和微生物的基因組的成本將輕松達到每年數十億美元。這些數據需要保存幾十年,甚至更長時間。
將數據壓縮顯然有助于解決其存儲問題。生物信息學專家已經使用像gzip這樣的標準壓縮工具將文件大小縮小到了原來的1/20。一些研究人員還使用針對基因組數據的更專業的壓縮工具,但這些工具并沒有被廣泛采用。我們兩個人都在研究數據壓縮算法,我們認為現在是時候提出一種效率更高、速度更快、更適合基因組數據獨特特性的新壓縮方案了。正如專用的視頻和音頻壓縮方案對于像YouTube和Netflix這樣的流媒體服務至關重要一樣,要從爆炸式增長的基因組數據中盡可能多地獲益,專門針對基因組數據的高效壓縮方案將是十分必要的。
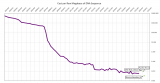
圖片來源:Stephens ZD,Lee SY,Faghri F,Campbell RH,Zhai C,Efron MJ,et al.2015,PLoS Biol 13(7).
人類基因組測序的增長:自2001年人類基因組序列草圖首次發表以來,測序的人類基因組數量和測序能力的增長速度都有了顯著提高。2015年后的三條線代表三種可能的增長曲線。
在我們解釋如何更好地壓縮基因組數據之前,讓我們仔細研究一下數據本身。“基因組”在這里指的是四種堿基核苷酸——腺嘌呤(adenine)、胞嘧啶(cytosine)、鳥嘌呤(guanine)和胸腺嘧啶(thymine)——的序列,它們分別由我們熟悉的DNA中的A、C、G、T四個字母表示。這些核苷酸出現在A-T和C-G堿基對組成的鏈中,人類基因組中的23對染色體都是由這兩種堿基對構成的。大多數人類細胞中,這些染色體包含約60億個核苷酸,包括編碼基因、非編碼元件(如染色體末端的端粒)、調節元件和線粒體DNA。Illumina、Oxford Nanopore Technologies和Pacific Biosciences等公司生產的DNA測序儀器,能夠在數小時內從一個人的DNA樣本中自動完成對其基因組的測序。
這些商業化的DNA測序儀不會產生整個基因組長度的ACGT字符串,而是產生大量子串或“讀數”(reads)。這些讀數會部分重疊,需要序列組裝軟件基于它們重建出完整的基因組。一般來說,當進行整個基因組測序時,每個基因組片段長度不超過100個讀數。
根據所使用的測序技術,讀數的長度可能從大約100到100,000個堿基對變化,讀數的總數可能從數百萬到數百億不等。短讀數可以發現單個堿基對突變,而較長的讀數更適用于檢測復雜的變異,如數千個堿基對的刪除或插入。
DNA測序是一個嘈雜的過程,讀數中包含錯誤是很常見的。因此,除了ACGT核苷酸字符串之外,每個讀數包含一個質量分數,表明測序儀對每個DNA核苷酸測序結果的信任度。測序儀將它們的質量分數表示為錯誤概率的對數。它們使用的算法是專有的,但事后可以檢查。如果質量得分為20(對應于1%的錯誤概率),用戶可以確認在已知的DNA序列中約1%的堿基對是不正確的。使用這些文件的程序依賴質量分數來將測序錯誤和突變區分開來。真正的突變會比測序錯誤顯示出更高的平均質量分數,也就是說其錯誤概率更低。
測序儀將字符串和質量分數以及一些其他元數據逐個讀數地粘在一起,形成所謂的FASTQ文件。一個完整基因組的FASTQ文件通常包含數十到數百千兆字節。
這些文件也非常冗余,這源于任何兩個人的基因組幾乎完全相同這個事實。平均而言,兩個人的基因組在每1,000個核苷酸中大約有一個核苷酸不同,通常這些基因差異是很有趣的。一些DNA測序針對特定的差異區域,例如,像23andMe這樣的DNA基因分型應用程序只尋找特定的變異,而刑事調查中的DNA分析則去尋找特定標記重復次數的變異。
但是,如果你不知道有趣的東西在哪里(比如當你試圖診斷一種未知基因來源的疾病時),你就需要對整個基因組進行測序,這就意味著你需要獲取更大量的測序數據。
測序數據的重復也來自于為清除錯誤而多次讀取基因組的相同部分。有時,一個樣本中包含一個序列的多個變異,因此你想重復對其進行測序以捕獲這些變異。比如說你正試圖檢測一個組織樣本中的一些癌細胞或一個孕婦的血液中的胎兒DNA痕跡,這可能就意味著要對每個DNA堿基對多次測序(通常超過100次)以區分罕見變異與更常見變異,以及它們與測序錯誤的真正區別。
讀數和參考基因組:一個DNA“讀數”(頂部字符串)與人的參考基因組的一小部分(底部字符串)大致匹配。插入、刪除和替換(由于DNA測序過程中的突變或噪聲)導致不完美匹配。為了編碼一個讀數,我們可以聲明其在參考基因組中的起始位置并描述所有變異。
現在,你應該更好地理解了為什么DNA測序會產生如此多的冗余數據。事實證明,這種冗余對于數據壓縮是有利的。對于相同的基因組數據塊,你可以只存儲一個副本,而不是存儲多個副本。
-
冗余
+關注
關注
1文章
112瀏覽量
20490 -
基因
+關注
關注
0文章
95瀏覽量
17455
原文標題:面臨挑戰的基因組數據壓縮技術(上)
文章出處:【微信號:IEEE_China,微信公眾號:IEEE電氣電子工程師】歡迎添加關注!文章轉載請注明出處。
發布評論請先 登錄
相關推薦





 你去將你的基因組測序了嗎?面臨挑戰的基因組數據壓縮技術
你去將你的基因組測序了嗎?面臨挑戰的基因組數據壓縮技術











評論