據悉,斯坦福大學等研究機構的研究人員建立了一個名為Tabula Muris的開源數據庫,收錄了小鼠的多個細胞類型及其基因表達數據。這項成果于上周發表在《Nature》雜志上。
研究者利用熒光活化細胞分選(FACS)方法和微流體液滴捕獲方法,從小鼠20個不同器官中分離出超過10萬個細胞,并開展單細胞RNA測序。這些數據讓人們能夠比較各個細胞類型和組織間的基因表達,從而更深入地了解細胞多樣性。
團隊成員以三只雌性小鼠和四只雄性小鼠為研究對象,利用FACS方法分離出44,949個細胞,并利用微流體方法分離出55,656個細胞。為了確定細胞類型,研究人員獨立分析每個器官,并使用聚類分析。
研究人員挑選出各個器官(包括腦、心臟、胰腺和胸腺等)的細胞,然后開展單細胞RNA測序,以獲取每個細胞的轉錄組。研究人員指出,FACS方法和微流體方法在大量基因表達譜上基本一致。
之后,研究人員又利用t-SNE算法來觀察各種器官的不同細胞類型之間的關系。他們發現,不同器官的細胞常常混在一起。他們還對每個組進行異質性評分,以評估它們是否由相關或不相關的細胞類型組成。
研究人員進一步發現,轉錄因子表達可以大致定義細胞類型。在用1,016個轉錄因子的平均表達來分類細胞時,他們發現此時產生的樹狀圖與利用所有基因產生的樹狀圖大體相似。當他們用細胞表面標志物或RNA剪接因子來重復此分析時,發現情況并非如此,表明轉錄因子能夠更好地確定細胞類型。
研究人員認為,這種細胞圖譜有助于細胞重編程。此外,小鼠的轉錄組圖譜也有助于發現新的細胞類型,揭開不同細胞類型的基因表達,并比較各個器官的細胞。
-
數據庫
+關注
關注
7文章
3904瀏覽量
65832
原文標題:Nature: 微流體液滴捕獲方法助力小鼠細胞的開源數據庫Tabula Muris
文章出處:【微信號:Microfluidics-Tech,微信公眾號:微流控科技】歡迎添加關注!文章轉載請注明出處。
發布評論請先 登錄
MySQL數據庫是什么
詳解《斯坦福 AI 報告 2025》:國產模型崛起、清華論文領先

斯坦福大學發布《2025 年人工智能指數報告》

阿里巴巴Qwen大模型助力開發低成本DeepSeek替代方案
Qwen大模型助力開發低成本AI推理方案
云數據庫是哪種數據庫類型?
斯坦福STANFORD FS725銣鐘
斯坦福研究:電動汽車電池實際壽命比預估長得多
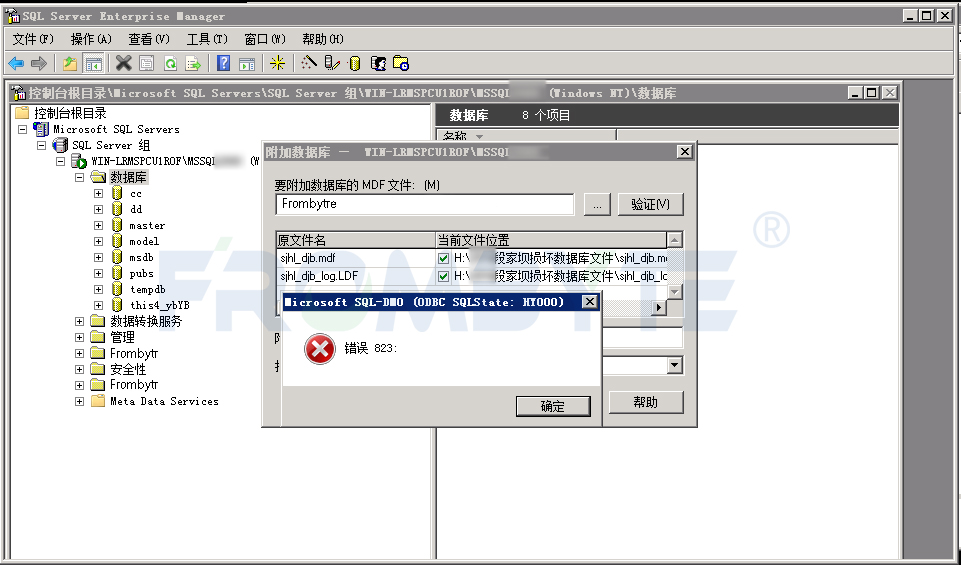
數據庫數據恢復—SQL Server數據庫出現823錯誤的數據恢復案例
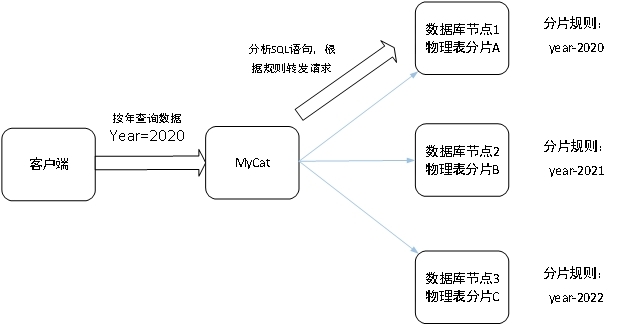
軟件系統數據庫的分庫分表設計





 斯坦福大學研究人員建立了一個名為Tabula Muris的開源數據庫
斯坦福大學研究人員建立了一個名為Tabula Muris的開源數據庫










評論